Splicing
La maturazione dell'mRNA: lo splicing
Negli eucarioti la trascrizione dell'RNA (acido ribonucleico) messaggero a partire da DNA grazie all'enzima RNA polimerasi DNA dipendente è seguito da tre processi di maturazione del messaggero (mRNA) prima che quest'ultimo possa essere tradotto.
Dalla trascrizione si ottiene quello che prende il nome di RNA trascritto primario che subisce:
- capping al 5';
- splicing;
- poliadenilazione al 3'.
In questa sede ci occuperemo di vedere più nel dettaglio il processo di splicing che permette la scissione delle sequenze non codificanti, gli introni, del trascritto primario ed il successivo legame delle sequenze codificanti, gli esoni, tra di loro in maniera consequenziale come si devono trovare nell'mRNA maturo.
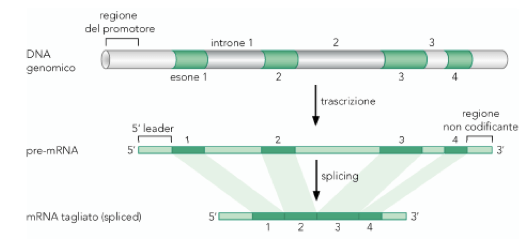
Schematizzazione del processo di splicing nel corso della maturazione dell'RNA messaggero.
Per la presenza di introni intervallati ad esoni codificanti, i geni eucarioti sono definiti "geni interrotti".
Lo splicing fu scoperto nel 1977 attraverso studi di ibridazione DNA-RNA nell'adenovirus, in particolare attraverso la comparazione della mappa di restrizione eseguita sul cDNA (DNA clonato, ottenuto in vitro per retrotrascrizione dell'mRNA) con quella ottenuta dal DNA genomico.

La comparazione del DNA genomico con il cDNA evidenzia l'esistenza di introni ed esoni.
Esistono due teorie per spiegare l'esistenza degli introni: nel modello precoce si ipotizza la perdita degli introni nei procarioti, in quello tardivo la comparsa di essi negli eucarioti.
Alcuni geni eucaristici, come ad esempio le immunoglobuline (Ig) caratterizzati da domini ripetuti, potrebbero essere il risultato della duplicazione e della successiva divergenza di esoni.
La biochimica dello splicing
Lo splicing avviene mediante due reazioni di esterificazione in cui si verifica l'attacco nucleofilo dell'OH (ossidrile) libero in 3' ad un legame fosfodiestere in 5'.
Le sequenze più conservate sono GU al 5' e AG al 3': teoricamente ogni sito di splicing al 5' potrebbe reagire con un altrettanto qualunque sito al 3', ma solitamente il processo di splicing avviene tra il 5' e il 3' dello stesso introne.
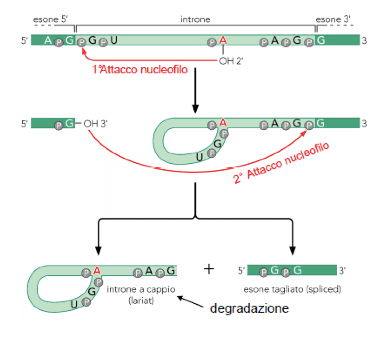
Gli eventi chimici alla base dell'escissione di un introne nel corso dello splicing.
Nel corso delle due reazioni di esterificazione non si ha consumo di energia in quanto l'ATP verrà consumata successivamente, nell'assemblaggio e nel funzionamento del complesso macchinario di splicing.

Struttura della giunzione che si forma nel corso del processo di splicing.
Lo spliceosoma
Il complesso di proteine ed RNA che permette lo splicing è detto spliceosoma. Gli RNA che costituiscono lo spliceosoma sono detti RNA small nuclear (snRNA), un singolo small nuclear RNA (100-300 bp) si associa a diverse proteine per formare quello che viene indicato con i termini "small ribonuclear protein (snRNP, pronunciato "snurp"), gli snRNP sono identificati con le seguenti sigle: U1; U2, U5, U4/6. Lo spliceosoma contiene in tutto circa 150 proteine.
Per il funzionamento dello spliceosoma e dunque per l'ottenimento dello splicing degli introni, sono fondamentali le interazioni RNA-RNA ed RNA-proteina.
Negli snRNA infatti esistono, ad esempio, delle regioni a singolo filamento (ricordiamo che gli RNA assumono conformazioni tridimensionali grazie a regioni complementari all'interno del filamento) complementari al 5' di splicing dell'introne, altre interazioni sono rappresentate nella figura sottostante:
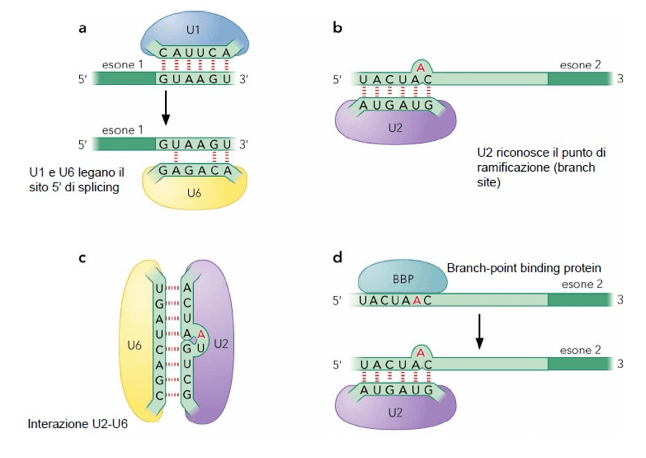
Interazioni RNA-RNA ed RNA-proteina nello spliceosoma.
Ed infine di seguito riportiamo con una schematizzazione le fasi della reazione di splicing attraverso cui un introne per volta (reazione tra l'OH in 3' e il 5' di splicing) viene tagliato, ripiegato, rimosso e le estremità dei due esoni che lo fiancheggiano sono infine legate tramite la seconda reazione di esterificazione promossa da U5. L'introne rimosso è degradato.
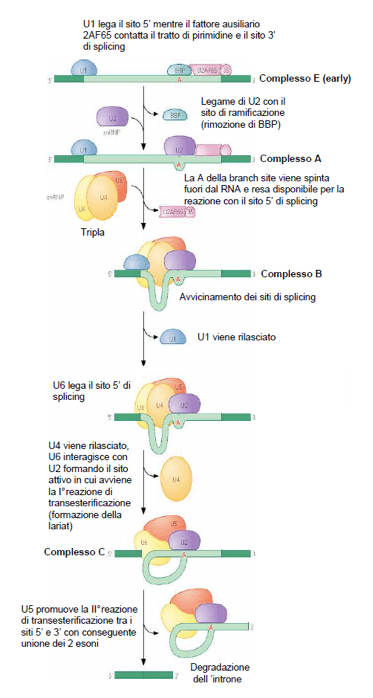
Schematizzazione delle reazioni di splicing.
Uno dei meccanismi di regolazione dell'espressione genica prevede l'utilizzo di quello che viene indicato come "splicing alternativo" ovvero si possono produrre mRNA maturi diversi a partire dallo stesso trascritto primario inserendo, ad esempio, un introne tagliato in un'altra "versione" della proteina.
Da processi di splicing diversi si otterranno proteine simili, con domini simili, ma diverse per regolazione e/o funzione.

Esempio di splicing alternativo: un trascritto primario dà luogo a due prodotti proteici.
Splicing e patologie
Lo splicing alternativo è importante anche nella patogenesi di alcune patologie, citiamo, ad esempio, l'atrofia muscolare spinale (SMA) nella quale la proteina SMN (proteina di sopravvivenza del motoneurone) è mutata e dunque si ha una progressiva e letale degenerazione dei motoneuroni con atrofia inizialmente dei muscoli degli arti inferiori e successivamente degli altri muscoli, sino a quelli respiratori.
Esistono due geni per SMN: smn1 e smn2, nella patologia smn1 è mutato e dunque non codifica per una proteina funzionante, mentre smn2 non riesce a sopperire alla funzionalità della proteina mancante poiché uno splicing alternativo spesso esclude l'esone 7 portando ad una versione non funzionante di smn2.
Lo splicing alternativo può riguardare poche copie della proteina smn2 con una prognosi migliore per i pazienti, oppure molte copie con prognosi peggiore.
Link correlati:
Quanto è lungo il DNA?
Che cosa è l'ereditarietà?
Che cos'è il il ciclo di Cori?
Le cellule nervose: il neurone
Che cosa sono i trasposoni?
Studia con noi